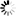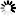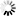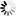Das Erbgut ist 30 Mal größer als das des Menschen und mehr als doppelt so groß wie das des bisherigen Rekordhalters: Ein internationales Team um den Konstanzer Evolutionsbiologen Axel Meyer und den Würzburger Biochemiker Manfred Schartl hat das größte bekannte Genom aller Tiere entschlüsselt: das Erbmaterial des Südamerikanischen Lungenfischs (Lepidosiren paradoxa). „Die Daten werden helfen zu ergründen, wie dem Vorfahren der heutigen Landwirbeltiere die Eroberung des Festlands gelang”, hieß es in einer Mitteilung zur Veröffentlichung der Studienergebnisse in der Fachzeitschrift „Nature”.
Lungenfische gelten als nächste heute noch vorkommende Verwandte jener Tiere, die sich vor rund 400 Millionen Jahren mit kräftigen Flossen an Land zogen - und dank ihrer Lungen dort überleben konnten. Auf sie gehen alle sogenannten Vierfüßer wie Menschen, andere Säugetiere, Amphibien, Reptilien und Vögel zurück. Von Lungenfischen gibt es den Angaben zufolge heute noch drei Linien: eine in Afrika, eine in Australien und eine in Südamerika, die unter anderem im Amazonasbecken vorkommt.
Von der Analyse des Erbguts erhoffen sich die Fachleute Erkenntnisse über die Ursprünge und weitere Entwicklung dieser ungewöhnlichen Tiere. „Die Evolution scheint sie vergessen zu haben”, schreiben die Forschenden. Denn diese uralten „lebenden Fossilien” sähen weitestgehend noch aus wie ihre Vorfahren im späten Devon vor grob 400 Millionen Jahren. Durch Vergleiche der Genomsequenzen lässt sich demnach etwa der Zusammenhang zwischen Flossenstrahlen der Knochenfische und Fingern von Landwirbeltieren belegen.
Rekord-Erbgut 30 Mal so groß wie das des Menschen
Dass die Genome der Lungenfische riesig sind, war bereits bekannt, doch die Studie zeigt nun, wie gigantisch sie tatsächlich sein können: Das Erbgut des Südamerikanischen Lungenfischs bestehe aus mehr als 90 Milliarden Basen, schreibt die Gruppe. Es sei damit 30 Mal so groß wie das Genom des Menschen und mehr als doppelt so groß wie das des bisherigen Rekordhalters, des Australischen Lungenfischs (Neoceratodus forsteri). „18 der 19 Chromosomen des Südamerikanischen Lungenfischs sind allein jeweils größer als das gesamte menschliche Genom”, wird Schartl in einer Mitteilung zur Studie zitiert.
Das liege an sogenannten autonomen Transposons. Diese DNA-Abschnitte „vermehren” sich und verändern dann ihre Position im Genom. Sie machten mehr als 90 Prozent des Lungenfisch-Genoms aus. Die Expansionsrate beim südamerikanischen Vertreter ist demnach mit Abstand die schnellste bekannte: Alle zehn Millionen Jahre sei sein Erbgut in der Vergangenheit um die Größe des gesamten menschlichen Genoms gewachsen.
Trotzdem sei das Lungenfisch-Genom überraschend stabil. Daher konnte das Forschungsteam nach eigenen Angaben aus den Sequenzen der heute noch lebenden Lungenfisch-Arten die ursprüngliche Architektur des Chromosomensatzes des ersten Vierfüßers rekonstruieren.
Vergleichsstudien ermöglichen Aufschlüsse über Evolution
Zudem lassen sich die Genome der verschiedenen Lungenfische vergleichen und so Rückschlüsse dazu ziehen, ob Unterschiede zwischen den noch heute lebenden Exemplaren auf genetische Ursachen zurückgehen. Der Australische Lungenfisch etwa hatte demnach noch Gliedmaßen-ähnliche Flossen, mit denen sich die Vorfahren einst an Land bewegen konnten. Bei den heutigen Lungenfischvertretern aus Afrika und Südamerika dagegen hätten sich diese Flossen, die im Knochenbau den menschlichen Armen und Beinen ähnelten, im Laufe der letzten rund 100 Millionen Jahre zu fadenförmigen Flossen zurückentwickelt.
Anhand der Analyse des Erbguts des Australischen Lungenfischs hatte das Team um Meyer und Schartl schon vor einigen Jahren gezeigt, dass gleiche Gene im Menschen und im Lungenfisch die Entwicklung der Lunge steuern. Die Lunge von Lungenfischen habe entwicklungsgeschichtlich die gleiche Herkunft wie die der Landwirbeltiere, einschließlich des Menschen. Auch seien Finger, Elle und Speiche in der Flosse des Tiers angelegt, wofür die gleichen Gene wie beim Menschen verantwortlich seien. An der aktuellen Studie arbeiteten auch Wissenschaftlerinnen und Wissenschaftler unter anderem aus Dresden, Hamburg, Schweden, Österreich und Frankreich mit.